Como a mineração de dados revelou padrões importantes em dados de células tumorais
E quando as análises estatísticas convencionais não apontam nenhuma diferença significativa entre dois conjuntos de dados? E se pequenas diferenças desencadeassem modificações estruturais em todo o restante dos dados?
Nesse post eu discuto um exemplo em que dois conjuntos de dados eram praticamente iguais, mas pequenas diferenças causaram um impacto estrutural significativo no restante dos dados. Embora as diferenças não fossem significativas, o movimento estrutural desencadeado por elas revelou informações importantes.
Existiam dois conjuntos, um obtido a partir de células normais e outro de células tumorais. Análises mais convencionais não apontavam nenhuma diferença significativa entre os conjuntos, afinal, os processos biológicos mais comuns estavam presentes em ambos.
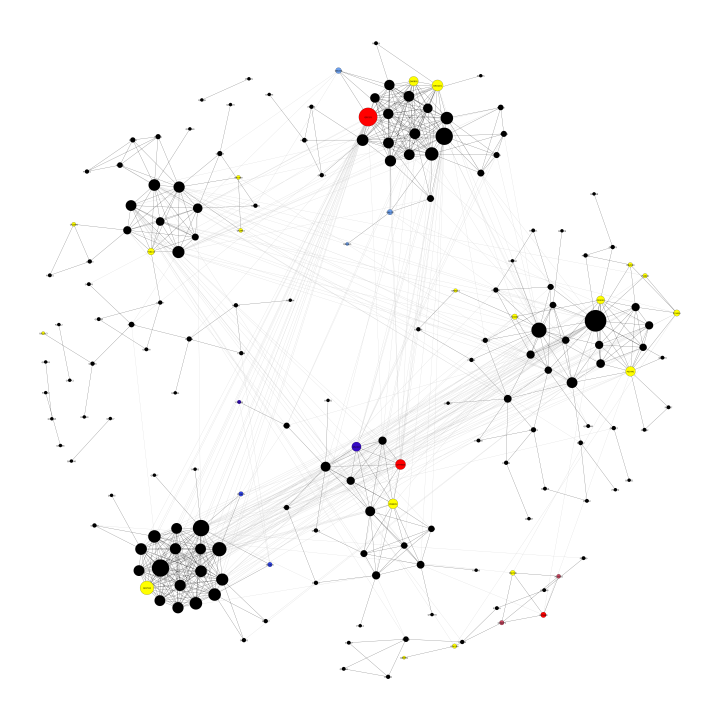
Eu transformei os conjuntos em redes, em que os nós representavam as linhas do conjunto original e as arestas representavam a existência e intensidade da conexão entre os elementos. Essa modelagem permitiu que eu aplicasse técnicas baseadas na teoria dos grafos, como detecção de comunidades, para identificar e analisar as diferenças de forma mais minuciosa.

Quando identifiquei e analisei as comunidades das redes os resultados começaram a ficar interessantes. Os nós de uma mesma comunidade estavam mais expressos nos mesmos processos biológicos, o que deixou bem claro o papel desses "grupos". Cada comunidade era formada por nós altamente envolvidos em processos biológicos essenciais, como respiratórios, reprodutivos, alimentação, crescimento, etc.
Outra ficou bem clara nesse momento: o "core" (nós na cor preta) dos grupos se mantinha intacto em ambas as redes. Nesse sentido, os nós de fora do core (os nós destacados mas cores amarela, vermelha, azul...) são aqueles que pertenciam somente a um dos dois conjuntos ou aos dois, mas em comunidades diferentes. Isso significa que os poucos elementos que existiam em apenas um dos conjuntos afetavam a estrutura da rede a ponto de "mover" nós de uma comunidade para a outra.
Essa modificação na estrutura da rede e a movimentação de nós entre as comunidades abriu um caminho promissor. Analisei essas modificações e seu efeito nas comunidades. E... bingo! A análise revelou uma expressiva reconfiguração no interactoma tumoral de proteínas secretadas com importantes papéis biológicos em doenças malignas, como o câncer. Essa reconfiguração revelou insights de como um tumor afeta os processos biológicos mais comuns de uma célula normal.
Essas análise e seus resultados deram origem a um artigo chamado "Community-based network analyses reveal emerging connectivity patterns of protein-protein interactions in murine melanoma secretome". Se você se interessou em saber os resultados dessas análises em um contexto biológico, clique aqui.
A conclusão que deixo é que analisar diferenças entre conjuntos de dados de forma estática pode ignorar os efeitos estruturais dessas diferenças no restante do conjunto. Isso acontece porque pequenas diferenças podem desencadear um movimento de reconfiguração estrutural no restante do conjunto, mesmo que ele seja igual em ambos os cenários em termos de dados e atributos.



